

차원 축소
> 차원축소는 매우 많은 피처로 구성된 다차원 데이터 세트의 차원을 축소해 새로운 차원의 데이터 세트를 생성하는 것입니다.
*차원의 저주
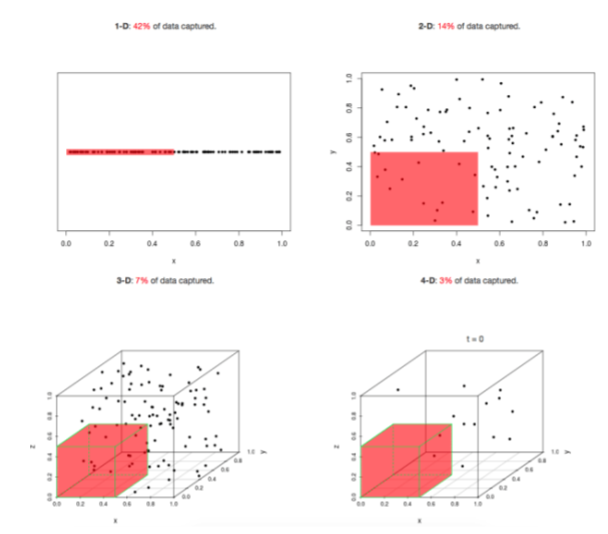
위 그림과 같이 차원이 증가할수록 해당 피처가 설명할수있는 비율이 감소합니다.
일반적으로 차원축소는 feature selection 과 feature extraction으로 나눌 수 있습니다.
feature selection은 특정 피처에 종속성이 강한 불필요한 피처를 삭제하는 개념.
fetaure extraction은 기존 피처를 저차원의 중요 피처로 압축해 추출하는 개념.
- PCA
PCA는 여려변수 간에 존재하는 상관관계를 이용해 이를 대표하는 주성분(Principal Component)을 추출해 차원을 축소하는 기법.
가장 높은 분산을 가지는 데이터의 축을 찾아 이축으로 차원을 축소하는데 이 축이 PCA의 주성분이 됩니다.
-실습
사이킷런의 붓꽃 데이터세트를 로딩한 뒤 보기 쉽게 DataFrame으로 변환합니다.
matplolib의 scatter를 이용해 sepal length , sepal width에 대한 각각의 붓꽃 품종의 산점도를 나타냅니다.
from sklearn.datasets import load_iris
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
# 사이킷런 내장 데이터 셋 API 호출
iris = load_iris()
# 넘파이 데이터 셋을 Pandas DataFrame으로 변환
columns = ['sepal_length','sepal_width','petal_length','petal_width']
irisDF = pd.DataFrame(iris.data , columns=columns)
irisDF['target']=iris.targetㅊ
irisDF.head(3)
setosa는 세모, versicolor는 네모, virginica는 동그라미로 표현했습니다. (marker)
markers=['^', 's', 'o']
for i, marker in enumerate(markers):
x_axis_data = irisDF[irisDF['target']==i]['sepal_length']
y_axis_data = irisDF[irisDF['target']==i]['sepal_width']
plt.scatter(x_axis_data, y_axis_data, marker=marker,label=iris.target_names[i])
plt.legend()
plt.xlabel('sepal length')
plt.ylabel('sepal width')
plt.show()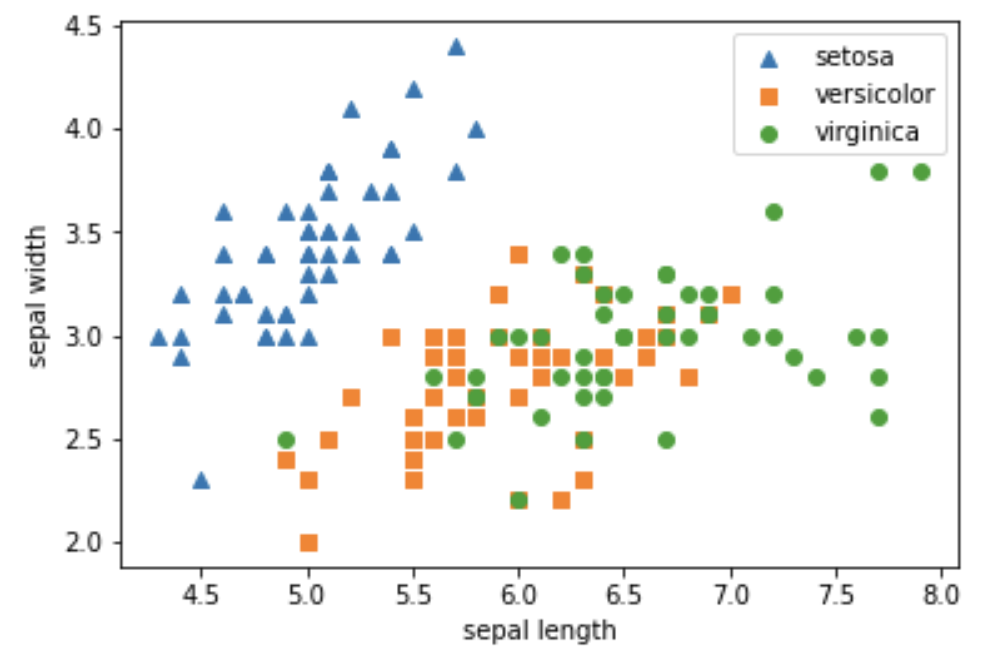
먼저 붓꽃 데이터 세트에 바로 PCA를 적용하기 전에 개별 속성을 함께 스케일링해야됩니다. PCA는 여려 속성의 값을 연산해야 하므로 속성의 스케일에 영향을 받기 때문입니다. (StandardScaler 이용)
from sklearn.preprocessing import StandardScaler
iris_scaled = StandardScaler().fit_transform(irisDF)
여기서 n_components는 PCA로 변환할 차원의 수를 의미합니다. 이후 fit와 transform을 이용해 PCA변환을 수행합니다.
새로운 2개의 속성으로 PCA변환된 데이터 세트를 시각화해보았습니다.
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
#fit(입력 데이터 세트) transform(입력 데이터 세트)를 호출해 PCA 변환.
pca.fit(iris_scaled)
iris_pca = pca.transform(iris_scaled)
iris_pca.shape
columns = [ 'pca_component_1','pca_component_2']
irisDF_pca = pd.DataFrame(iris_pca,columns=columns)
irisDF_pca['target'] = iris.target
irisDF_pca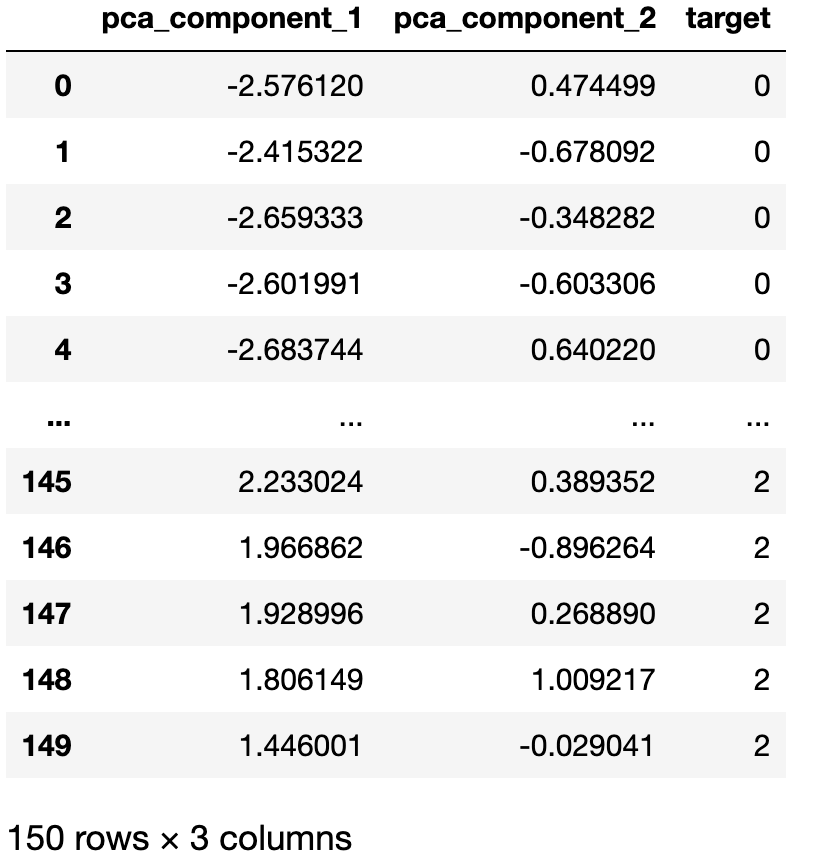
PCA변환후의 데이터를 바탕으로 처음 했던것과 같이 각 품종에 따른 산점도를 그려보았습니다.
columns = [ 'pca_component_1','pca_component_2']
irisDF_pca = pd.DataFrame(iris_pca,columns=columns)
irisDF_pca['target'] = iris.target
markers = ['^','s','o']
for i , marker in enumerate (markers):
x_axis_data = irisDF_pca[irisDF_pca['target'] == i]['pca_component_1']
y_axis_data = irisDF_pca[irisDF_pca['target'] == i]['pca_component_2']
plt.scatter(x_axis_data,y_axis_data,marker=marker,label=iris.target_names[i])
plt.legend()
plt.xlabel('pca_component_1')
plt.ylabel('pca_component_2')
plt.show()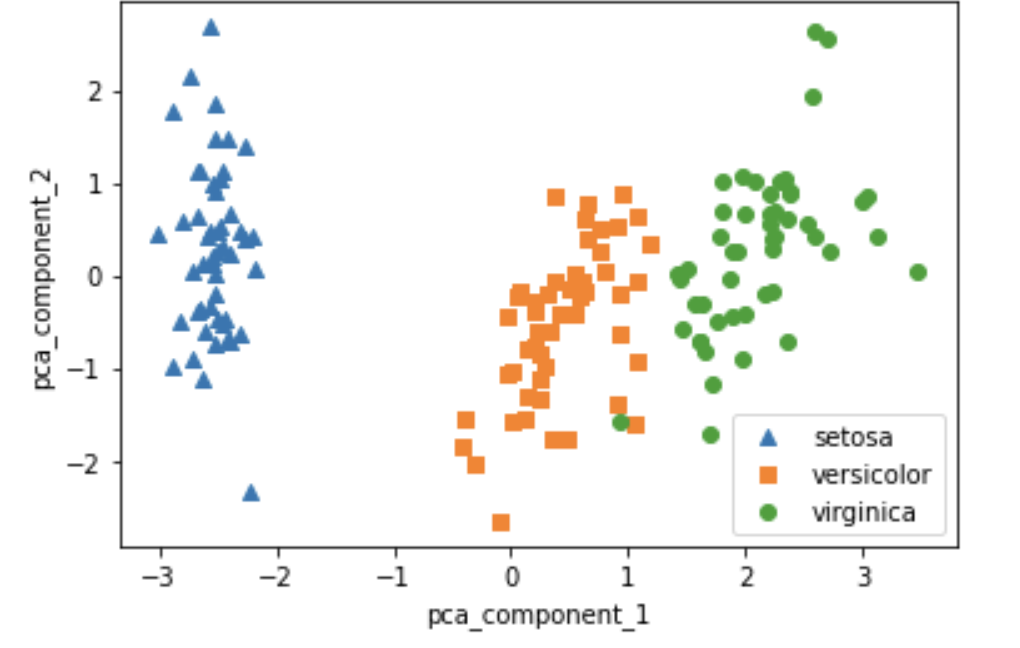
PCA 변환으로 이루어진 2개의 속성은 얼만큼 잘 설명하고 있을까요?
이때 쓰이는 메소드가 explained_variance_ratio_ 입니다.!
pca.explained_variance_ratio_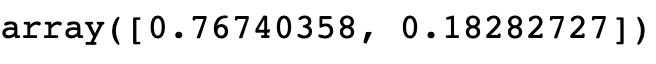
다음으로는 좀 더 많은 피처를 가진 데이터 세트를 적은 PCA 컴포넌트 기반으로 변환한 뒤, 예측영향도가 어떻게 변환 되지는 알아보겠습니다.
import pandas as pd
df = pd.read_excel('credit_card.xls', sheet_name='Data',header=1)
print(df.shape)
df.head(3)
몇몇 속성들의 이름을 알기 쉽게 rename 해줍시다.
df.rename(columns={'PAY:0':'PAY_1','default payment next month':'default'},inplace=True)
y_target = df['default']
X_features = df.drop('default',axis=1)
해당 데이터 세트는 23개의 속성 데이터가 존재하므로 각 속성끼리 상관도가 매우 높을 것입니다. corr()메소드를 이용해 각 속성 간의 상관도를 구한 뒤 이를 Seaborn의 heatmap으로 시각화 해보겠습니다.
import seaborn as sns
import matplotlib.pyplot as plt
corr = X_features.corr()
plt.figure(figsize=(14,14))
sns.heatmap(corr,annot=True,fmt='.1g')
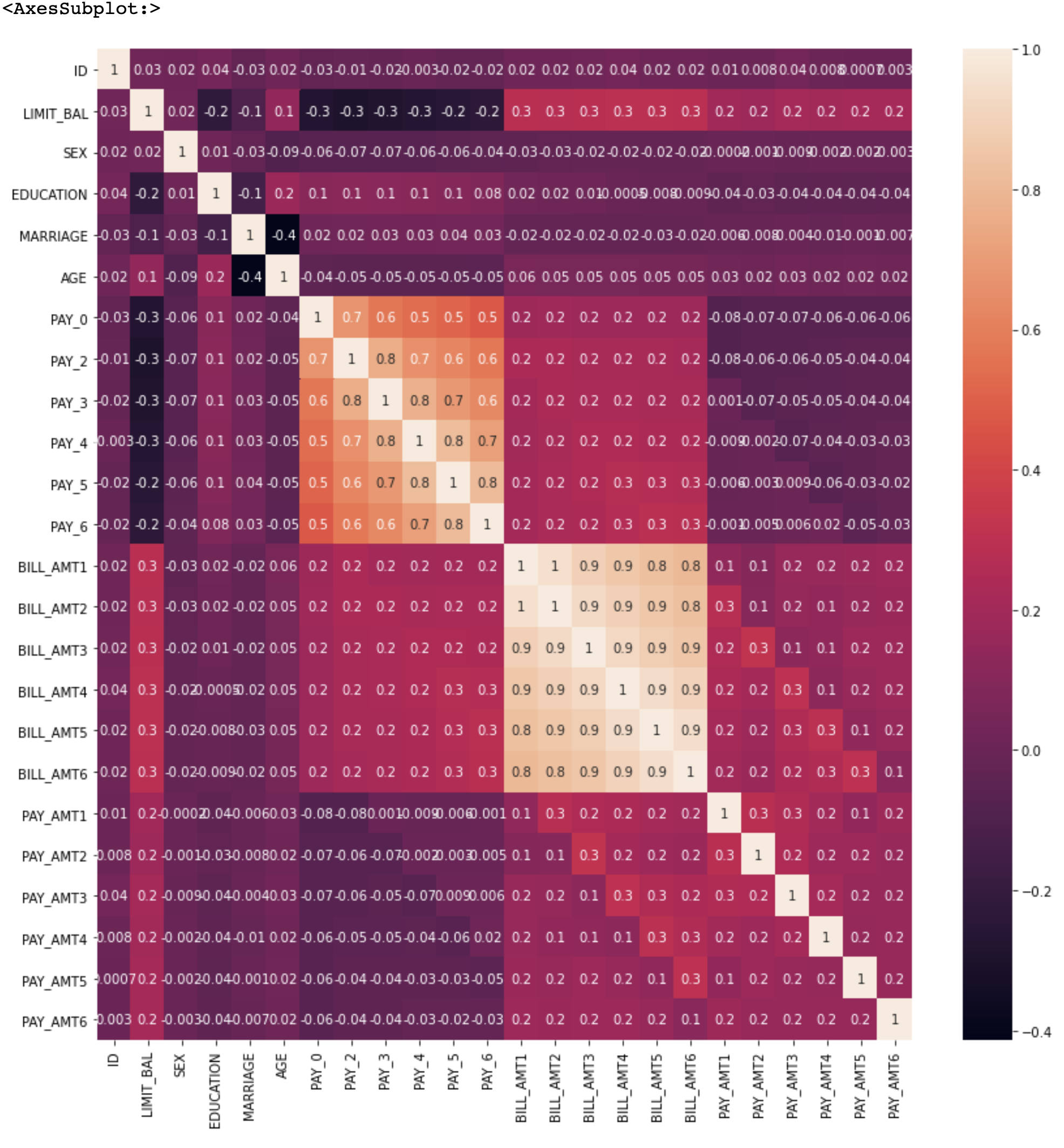
BILL_AMT1 ~ BLL_AMT6의 속성이 0.9이상 또한 PAY_1~PAY_6까지의 속성도 상관도가 높은걸 알 수 있습니다.
이렇게 높은 상관도를 가진 속성들은 소수의 PCA만으로도 자연스럽게 이 속성들의 변동성을 수용할 수 있습니다.
BILL_AMT1 ~ BLL_AMT6까지 6개의 속성을 2개의 컴포넌트로 PCA변환을 수행해 보겠습니다.
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
#BILL_AMT1 ~ BILL_AMT6까지 6개의 속성명 생성.
cols_bill = ['BILL_AMT' + str(i) for i in range(1,7)]
print("대상 속성명 :",cols_bill)
#2개의 PCA속성을 가진 PCA객체 생성하고 , explained_variance_ratio_계산을 위해 fit()호출
scaler = StandardScaler()
df_cols_scaled = scaler.fit_transform(X_features[cols_bill])
pca = PCA(n_components=2)
pca.fit(df_cols_scaled)
print("PCA Component별 변동성:", pca.explained_variance_ratio_)
2개의 PCA 컴포넌트만으로도 6개의 속성의 변동성을 95%이상 설명할 수 있습니다.
원본 데이터 세트와 6개의 컴포넌트로 PCA변환한 데이터 세트의 분류 예측 결과를 상호 비교해보겠습니다.
먼저 랜덤포레스트를 이용해 예측해보겠습니다.
import numpy as np
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score
rcf = RandomForestClassifier(n_estimators=300,random_state=777)
scores = cross_val_score(rcf,X_features,y_target,scoring='accuracy',cv=3)
print('CV=3 인 경우의 개별 Fold세트별 정확도',scores)
print('평균 정확도 : {:.3f}'.format(np.mean(scores)))
이번에는 원본 데이터 세트와 6개의 컴포넌트로 PCA변환한 데이터 세트의 분류 예측 결과를 상호 비교해보겠습니다.
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
df_scaled = scaler.fit_transform(X_features)
pca = PCA(n_components=6)
df_pca = pca.fit_transform(df_scaled)
scores_pca = cross_val_score(rcf,df_pca,y_target,scoring='accuracy',cv=3)
print("CV=3 인 경우의 PCA 변환된 개별 Fold 세트별 정확도",scores_pca)
print("PCA 변환 데이터 세트 평균 정확도 :",np.mean(scores_pca))

-LDA (Linear Discriminant Analysis)
LDA는 선형 판별 분석법으로 불리면 PCA와 매우 유사합니다. LDA는 PCA와 유사하게 입력 데이터 세트를 저차원 공간에 투영해 차원을 축소하는 기법이지만, 중요한 차이는 LDA는 지도학습의(분류)에서 사용하기 쉽도록 개별 클래스를 분별할 수 있는 기준을 최대한 유지하면서 차원을 축소합니다.
-실습
LDA 변환할때 가장 유의해야 할점은 LDA는 PCA와 다르게 비지도학습이 아닌 지도학습입니다. 그러므로 클래스의 결정값이 변환시에 필요합니다. lda객체에 fit()메소드를 호출할 때 결정값이 iris.target이 입력되야 합니다.!!
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from sklearn.preprocessing import StandardScaler
from sklearn.datasets import load_iris
iris = load_iris()
iris_scaled = StandardScaler().fit_transform(iris.data)
lda = LinearDiscriminantAnalysis(n_components=2)
lda.fit(iris_scaled,iris.target)
iris_lda = lda.transform(iris_scaled)
print(iris_lda.shape)
LDA변환 후 각 품종의 산점도를 나타내었습니다.
import pandas as pd
import matplotlib.pyplot as plt
lda_columns = ['lda_component_1','lda_component_2']
irisDF_lda = pd.DataFrame(iris_lda , columns = lda_columns)
irisDF_lda['target'] = iris.target
markers=[ '^','s','o']
for i,marker in enumerate(markers):
x_axis_data = irisDF_lda[irisDF_lda['target'] == i]['lda_component_1']
y_axis_data = irisDF_lda[irisDF_lda['target'] == i]['lda_component_2']
plt.scatter(x_axis_data,y_axis_data,marker=marker,label=iris.feature_names[i])
plt.legend()
plt.xlabel('lda_component_1')
plt.ylabel('lda_component_2')
plt.show()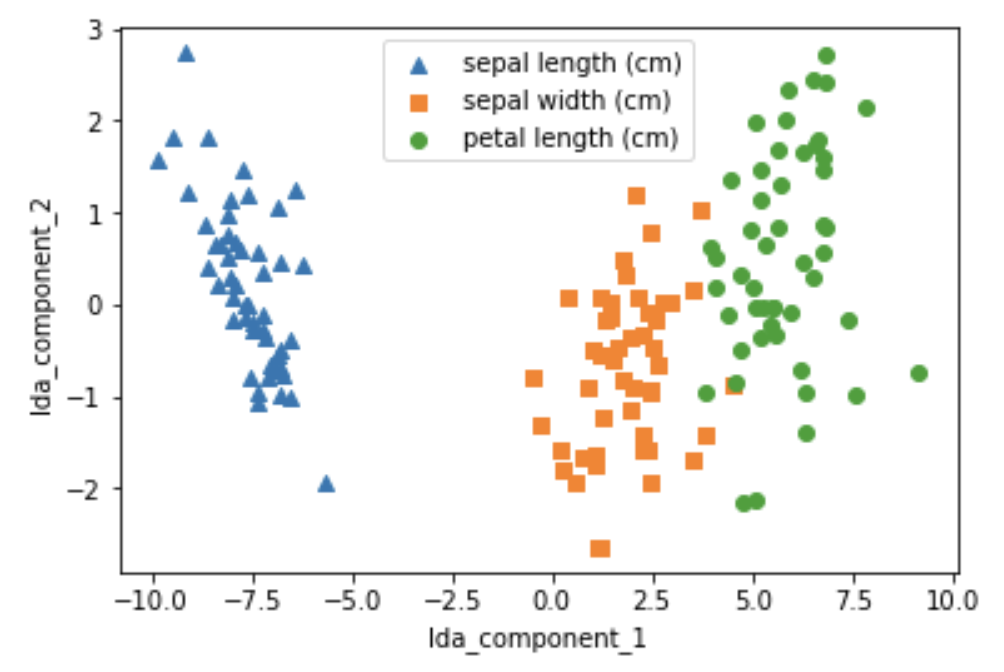
-SVD(Singular Value Decomposition)
SVD 역시 PCA와 유사한 행렬 분해 기법을 이용합니다. PCA의 경우 정방행렬만을 고유벡터로 분해할 수 있지만 SVD는 정방행렬뿐 아니라 비정방행렬에서도 적용할 수 있습니다.
-실습
import numpy as np
from numpy.linalg import svd
#4 4 랜덤 행렬 생성.
np.random.seed(777)
a = np.random.randn(4,4)
print(np.round(a,3))
SVD를 적용해 U , Sigma V**t 를 도출합니다.
U , Sigma , Vt = svd(a)
print(U.shape,Sigma.shape,Vt.shape)
print("U matrix:\n",np.round(U,3))
print("Sigma Value:\n",np.round(Sigma,3))
print("V transpose matrix:\n",np.round(Vt,3))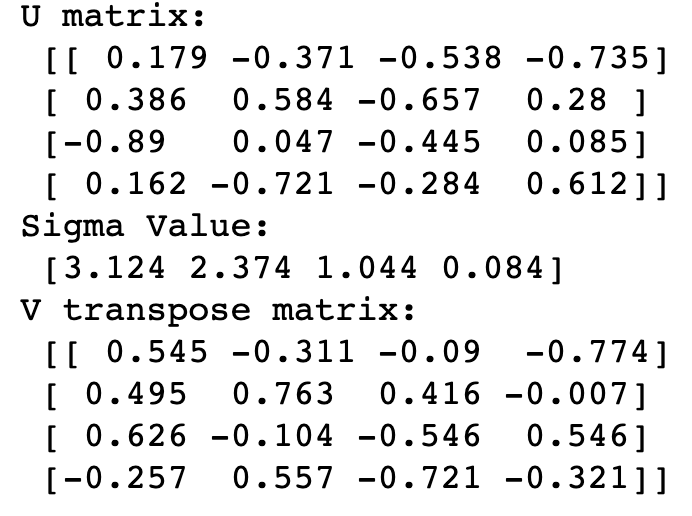
Truncated SVD
> Sigma에 있는 대각원소, 즉 특이값 중 상위 일부 데이터만 추출해 분해하는 방식
from sklearn.decomposition import TruncatedSVD, PCA
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
iris = load_iris()
iris_ftrs = iris.data
tsvd = TruncatedSVD(n_components=2)
tsvd.fit(iris_ftrs)
iris_tsvd = tsvd.transform(iris_ftrs)
plt.scatter(x=iris_tsvd[:,0],y=iris_tsvd[:,1],c=iris.target)
plt.xlabel("TruncatedSVD Component 1")
plt.ylabel("TruncatedSVD Component 2")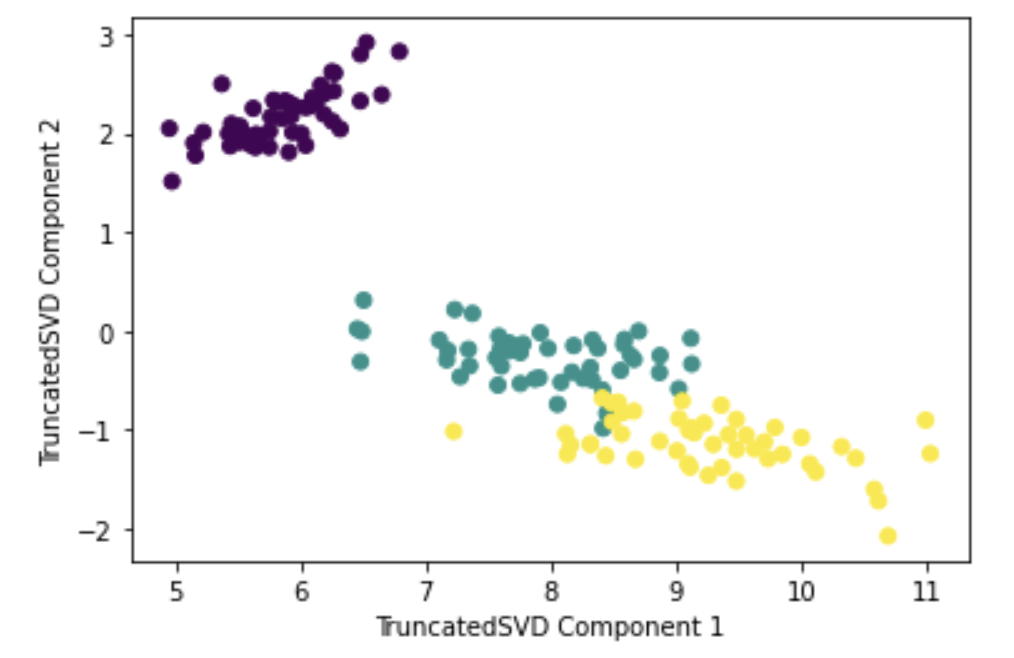
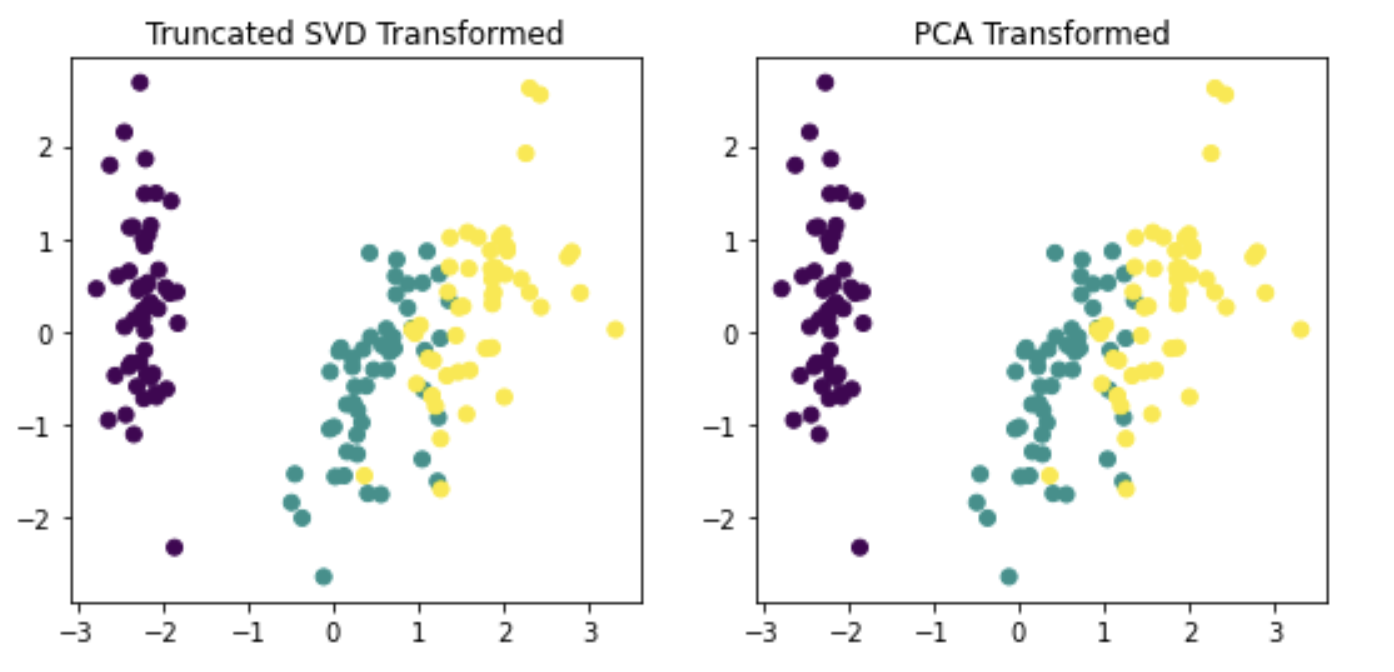
PCA와 TruncatedSVD 클래스 구현을 보면 두개 모두 SVD를 이용해 행렬분해합니다.
> 붗꽃데이터를 스케일링으로 변환한 뒤에 PCA , TruncatedSVD 클래스 변환을 해보면 거의 동일한 것을 알 수있습니다.
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
iris_scaled = scaler.fit_transform(iris_ftrs)
tsvd = TruncatedSVD(n_components=2)
tsvd.fit(iris_scaled)
iris_tsvd = tsvd.transform(iris_scaled)
pca = PCA(n_components=2)
pca.fit(iris_scaled)
iris_pca = pca.transform(iris_scaled)
fig , (ax1,ax2) = plt.subplots(figsize=(9,4),ncols=2)
ax1.scatter(x=iris_tsvd[:,0],y=iris_tsvd[:,1],c=iris.target)
ax2.scatter(x=iris_pca[:,0],y=iris_pca[:,1],c=iris.target)
ax1.set_title('Truncated SVD Transformed')
ax2.set_title('PCA Transformed')
-NMF(Non-Negative Matrix Factorization)
NMF는 Truncated SVD와 같이 낮은 랭크를 통한 행렬근사방식의 변형
from sklearn.decomposition import NMF
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
%matplotlib inline
iris = load_iris()
iris_ftrs = iris.data
nmf = NMF(n_components=2)
nmf.fit(iris_ftrs)
iris_nmf = nmf.transform(iris_ftrs)
plt.scatter(x=iris_nmf[:,0], y= iris_nmf[:,1], c= iris.target)
plt.xlabel('NMF Component 1')
plt.ylabel('NMF Component 2')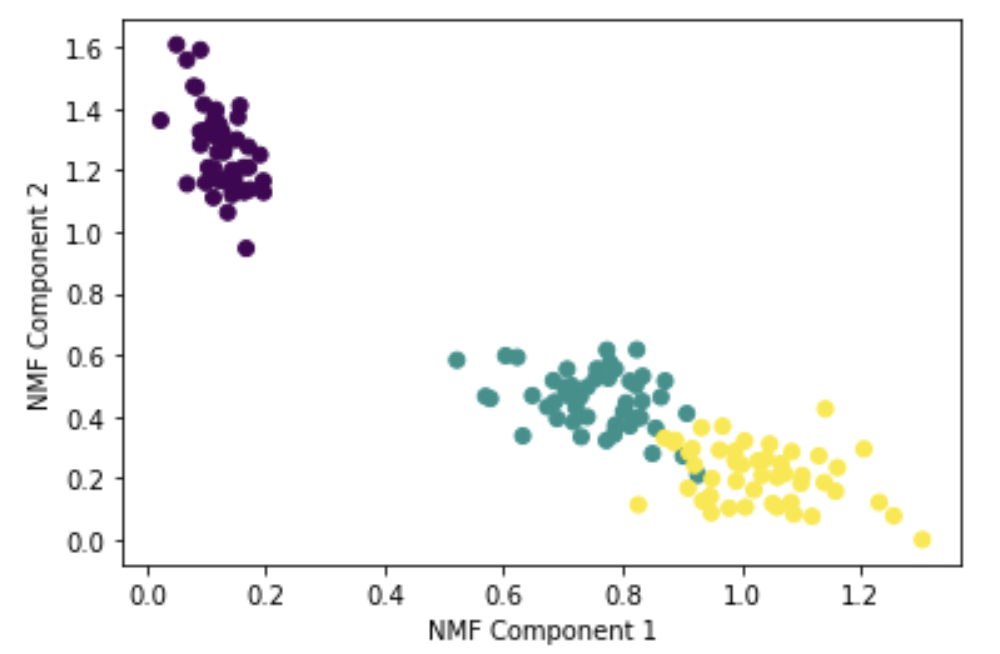
'DNN > 머신러닝' 카테고리의 다른 글
| 성능지표 / 평가 (0) | 2021.06.25 |
|---|---|
| 사이킷런으로 타이타닉 생존자 예측 (0) | 2021.06.23 |
| 붓꽃 품종 예측 , 사이킷런 , Model Selection , 데이터 전처리 (0) | 2021.06.23 |
| Pandas (0) | 2021.06.22 |
| Numpy(넘파이) (0) | 2021.06.22 |

포스팅이 좋았다면 "좋아요❤️" 또는 "구독👍🏻" 해주세요!



